
在#癌症#早期筛查中,基于外周血cfDNA的液体活检是一种极具潜力的检测手段:几乎所有肿瘤释放的cfDNA均可能在外周血检出,其所携带的突变、甲基化等多组学标志物在肿瘤的液体活检中具有重要诊断价值。有研究表明,联合检测肿瘤的突变、甲基化等多组学标志物可以显著提高癌症检测的效果【1】。当前大多数cfDNA检测技术仅能分析单个组学的标志物,而一次抽血提取到的cfDNA通常只够做一次检测。因此,多数既往cfDNA研究只报道了单个组学的标志物,难以比较不同类型的标志物在不同研究、不同队列中的表现,也难以结合多种标志物来开发更优的癌症检测方法。

近日,国家癌症中心/中国医学科学院肿瘤医院焦宇辰课题组、赵宏课题组和曲春枫课题组合作在Science Translational Medicine发表了题为 Simultaneous analysis of mutations and methylations in circulating cell-free DNA for hepatocellular carcinoma detection 的研究论文。研究团队开发了一种名为MCP(Mutation Capsule Plus)的方法,该方法可在不降低灵敏度的前提下对一份cfDNA样本实施多重分析,包括同时检测基因突变和甲基化信息,以及在全基因组范围内发现新的甲基化标志物,这一技术非常适用于以cfDNA为对象的癌症液体活检研究。进而,研究团队将这项技术应用于肝癌检测,通过MCP技术筛选了肝癌特异性的高甲基化标志物,与突变标志物组合构建了肝癌检测模型,通过独立验证集的验证表明,该模型能很好的区分肝癌与非肝癌个体。随后,通过纳入311例经甲胎蛋白(AFP)联合 B超(US)检测阴性的HBV携带者的前瞻队列,进一步证实了基于MCP技术建立的肝癌检测模型在肝癌早筛中的优良表现。总之,MCP方法能够识别cfDNA中的突变和甲基化两种不同类型的肿瘤标志物,可使肿瘤早期筛查更精准。通过这一小规模的肝癌筛查案例的表现,表明未来有望在更多癌种中发现新的标志物并开发相应的液体活检早筛方案,用于更大规模的研究。

文章发表在Science Translational Medicine上
引言
多个已有的肿瘤液体活检研究已证实,通过检测cfDNA突变可有效识别肿瘤患者【2-3】。然而,对于cfDNA产量较低的样本,精准检出低频突变比较困难;另外,部分肿瘤可能并不带有特征性驱动基因突变,也就无从通过cfDNA进行突变检测。因此,仅进行突变检测可能会造成一定的假阴性结果【4】。DNA甲基化是癌症进展中的早期事件,所有癌症类型中几乎均有异常的DNA甲基化。通过检测cfDNA甲基化对癌症进行诊断及监测也成为一种有效的无创活检方案【5-6】。
多组学标志物的联合检测,如对粪便标本中突变,甲基化和蛋白标志物的联合检测,可以显著提高癌症检测的效果【1】。然而,单次抽血提取到的cfDNA产量通常较低,只能支持单次检测;而大多数cfDNA检测技术只能检测到一种类型的标志物【7】。因此,目前以cfDNA为对象的研究多局限于单个组学的标志物(如突变或甲基化),很难对来源于不同研究、不同队列的突变和甲基化数据进行比较,也无法通过组合形成多组学标志物联合检测方案。为了解决该问题,研究团队开发出了MCP (Mutation Capsule Plus) 技术,该技术不仅能在一个反应中同时检测突变和甲基化,还可以实现甲基化标志物的从头发现。此外,MCP技术的另一个特点体现在其可以支持对同一份cfDNA样本的多次检测,有效提高了cfDNA样本的利用率。这些优势都使得MCP技术非常适用于以cfDNA为对象的癌症液体活检研究。作为对MCP技术应用于真实临床样本的考验,研究团队使用该技术建立了肝癌检测模型,该模型在多个独立验证队列中展示出较好的性能。
主要研究结果
1. MCP技术的建立和性能验证
为同时保留突变和甲基化信息,该团队放弃传统的重亚硫酸氢盐转化,而选用甲基化敏感型限制性内切酶对cfDNA进行处理,将GCGC酶切位点的甲基化状态转化为该位点的切割状态,即甲基化的cfDNA被完整保留而非甲基化的cfDNA在酶切位点处发生切割。随后,每个cfDNA片段都被连接独立标签识别序列(UID)并构建文库(pre-MCP文库),该pre-MCP文库可支持多次检测,通过对目标基因和甲基化位点的靶向扩增、高通量测序和生信分析即可解析cfDNA样本所携带的突变和甲基化改变。此外,使用一组特殊设计的引物,还可以在全基因组水平筛选高甲基化的GCGC酶切位点,从而发现新的甲基化标志物。
研究团队首先验证了MCP技术检测突变的性能。采取的方法是对不同DNA起始量下,不同突变频率的标准品分别进行检测。结果显示,40ng DNA起始量下,MCP对于0.1%, 0.05%, 0.025%突变频率下的检测灵敏度分别为100%,95%和73.8%,特异性为100%。此外,MCP在更低起始量下的低频突变检测中也表现出较好的性能:当分别以5ng、10ng和20ng DNA起始时,对于频率为0.1%的突变,检测灵敏度分别为67.5%、93.8%和100%。MCP技术能够检测多种变异类型,除了常见的单核苷酸突变(SNV)、小片段插入缺失(INDEL)外,还能检测融合突变、病毒基因组整合位点等。
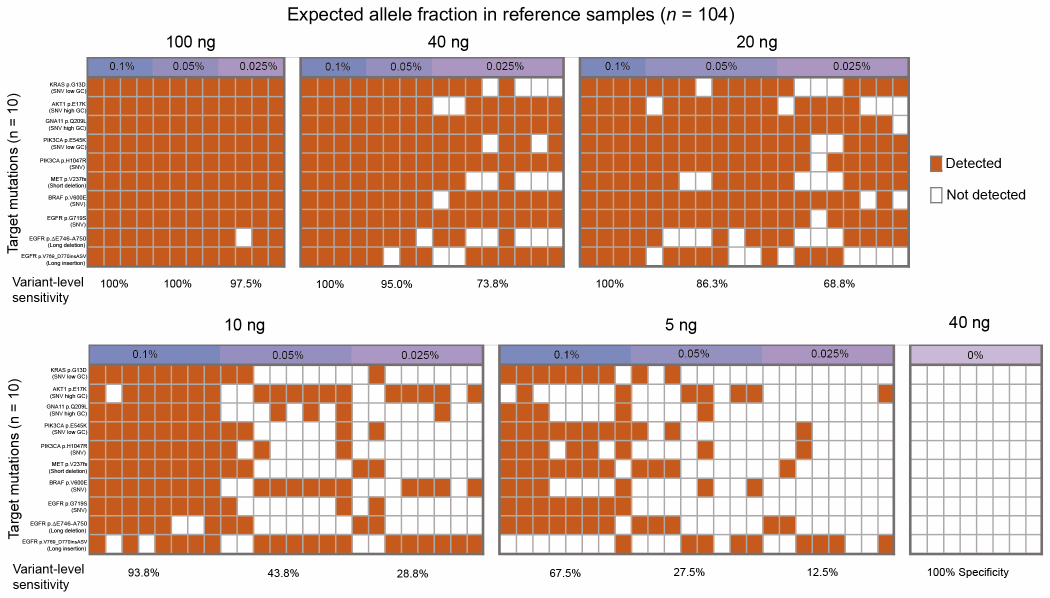
图1 MCP技术检测突变的性能
研究团队接着验证了MCP技术检测甲基化的性能。通过对一系列不同甲基化频率的标准品(25%, 10%, 1%, 0%)进行检测,结果显示,MCP技术检测到的甲基化频率与理论甲基化频率之间保持较高的一致性,能检测到频率低至1%的甲基化。作者还将MCP技术与两种常见的基于重亚硫酸氢盐转化的甲基化检测方法进行了比较,结果进一步证实MCP技术检测甲基化的可靠性。
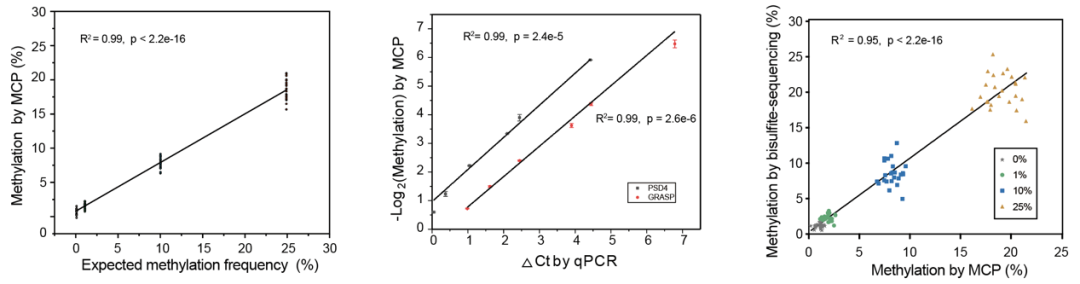
图2 MCP技术检测甲基化的性能
2. 利用MCP技术建立肝癌检测模型
为了验证MCP技术在临床样本中的实际效果,研究团队将该技术用于肝癌无创活检试剂盒的开发。两种类型的标志物被用于肝癌检测模型的构建:1)肝癌特异性高甲基化标志物;2)肝癌高频发生的基因突变,包括TP53, CTNNB1, TERT启动子突变以及HBV整合断点。
首先,在30例肝癌和30例非肝癌个体中筛选了肝癌特异性的高甲基化标志物,结合之前研究报道的具有肝癌诊断价值的甲基化标志物,在一组训练集中(60例肝癌和60例非肝癌)通过随机森林算法筛选出了10个表现优异的甲基化位点。最终联合突变标志物构建了突变-甲基化联合检测肝癌的模型。该模型在训练集中的灵敏度为93%, 特异性为95%。在一组独立的验证集(58例HCC和198例non-HCC)中,该模型表现出90%的灵敏度和94%的特异性。在训练集和验证集样本中,该模型对于不同阶段肝癌的检测灵敏度表现为,0期:86%,A期:91%,B期:93%,C期:100%,所有阶段的灵敏度为92%,特异性为95%。在上述样本中AFP对于0期,A期,B期,C期及所有阶段肝癌的诊断灵敏度分别为43%,53%,61%,25%和53%。

图3 基于MCP技术构建的肝癌检测模型的表现
3. 将肝癌检测模型应用于前瞻队列
研究团队将此模型应用于311例甲胎蛋白(AFP)/B超(US)阴性的无症状HBV携带者,进一步验证了此模型在前瞻队列肝癌早筛中的表现。模型预测结果显示共22例样本为阳性,其中4例确诊为肝癌,而检测阴性的289例样本里仅有1例确诊为肝癌。结果表明,该模型在前瞻队列中实现了80%的灵敏度,94%的特异度和18%的阳性预测值。上述结果提示了该cfDNA多组学无创活检方案具有从无症状前瞻队列中检测出容易被传统筛查方案(AFP/US)漏检的早期肝癌的能力。

结语
本研究报道了一种新的cfDNA检测技术-MCP,该技术可以在一份cfDNA样本中同时检测突变和甲基化,提高了珍贵的cfDNA样本的利用效率,体现出在cfDNA液体活检领域的一定优势。研究团队利用MCP技术构建了突变-甲基化联合检测肝癌模型,其在验证集中展现出较好的性能。最后,研究团队在无症状HBV携带者前瞻队列中进一步证实了该模型的检测效能,展示了其发现容易被常规筛查手段漏掉的早期肝癌的潜力。
这些研究结果表明了MCP作为一种高效能的cfDNA多组学标志物联合检测技术,在肝癌检测领域展现出广阔的临床应用前景。未来有望利用MCP技术在更多癌种发现新标志物以及开发无创活检产品,并进一步实现多个癌种的联合检测。
国家癌症中心/中国医学科学院肿瘤医院焦宇辰课题组有博士后岗位开放,诚挚欢迎有生物信息学、基因编辑及协同致死筛选、类器官等背景的优秀博士毕业生加入,共同挑战重大科学及转化问题。
课题组介绍:http://pumc.teacher.360eol.com/teacherBasic/preview?teacherId=4536
简历投递(有意者请将个人简历等材料发至):
https://jinshuju.net/f/ZqXwZt
原文链接:
http://www.science.org/doi/10.1126/scitranslmed.abp8704
参考文献
1. T. F. Imperiale, D. F. Ransohoff, S. H. Itzkowitz, T. R. Levin, P. Lavin, G. P. Lidgard, D. A. Ahlquist, B. M. Berger, Multitarget stool DNA testing for colorectal-cancer screening. New England Journal of Medicine370, 1287-1297 (2014).
2. J. D. Cohen, L. Li, Y. Wang, C. Thoburn, B. Afsari, L. Danilova, C. Douville, A. A. Javed, F. Wong, A. Mattox, Detection and localization of surgically resectable cancers with a multi-analyte blood test. Science359, 926-930 (2018).
3. Lennon, A.M., Buchanan, A.H., Kinde, I., Warren, A., Honushefsky, A., Cohain, A.T., Ledbetter, D.H., Sanfilippo, F., Sheridan, K., and Rosica, D. Feasibility of blood testing combined with PET-CT to screen for cancer and guide intervention. Science 369, eabb9601 (2020).
4. A. M. Aravanis, M. Lee, R. D. Klausner, Next-generation sequencing of circulating tumor DNA for early cancer detection. Cell168, 571-574 (2017).
5. J.-P. Issa, CpG island methylator phenotype in cancer. Nature Reviews Cancer4, 988-993 (2004).
6. H. Schwarzenbach, D. S. Hoon, K. Pantel, Cell-free nucleic acids as biomarkers in cancer patients. Nature Reviews Cancer11, 426-437 (2011).
7. T. F. Imperiale, D. F. Ransohoff, S. H. Itzkowitz, T. R. Levin, P. Lavin, G. P. Lidgard, D. A. Ahlquist, B. M. Berger, Multitarget stool DNA testing for colorectal-cancer screening. New England Journal of Medicine370, 1287-1297 (2014).


想了解更多精彩内容,快来关注BioArt生物艺术
| 留言与评论(共有 0 条评论) “” |