
撰文丨十一月
神经多样性的遗传学编码机制一直是神经领域的兵家必争之地。神经元是迄今为止动物中多样性最大的一种。黑腹果蝇的大脑为该问题的解决提供了一个良好的遗传学模型,其中视神经叶组成了果蝇大脑的三分之二,易于进行遗传操作且尺寸较小,可以用于神经网络的遗传编码的研究。以此为工作模型,美国纽约大学Claude Desplan研究组、Richard Bonneau研究组以及Mehmet Neset Ozel(第一作者)合作在Science发文题为Coordinated control of neuronal differentiation and wiring by sustained transcription factors,果蝇视觉神经系统的200个神经元可以被10个广泛表达的转录因子的排列组合进行解码,为有丝分裂后的神经元细胞命运特异化提供了框架。

视神经叶的神经网络主要由椎板(Lamina)、髓质(Medulla)、小叶(Lobula)以及小叶板(Lobula plate)组成(图1)。视神经叶被分为800个神经元,与果蝇最终的复眼数量相同。因为视神经组成具有重复的神经通路,作者们的实验室曾经进行过一个大规模视神经叶的单细胞RNA-seq,覆盖了从早期蛹到成年的6个不同的时间点【1】。所有神经元的群组是由各种不同神经元组成并且具有不同的形态结构,因此可以用不同神经元的表达网络特征回溯不同的遗传编码。
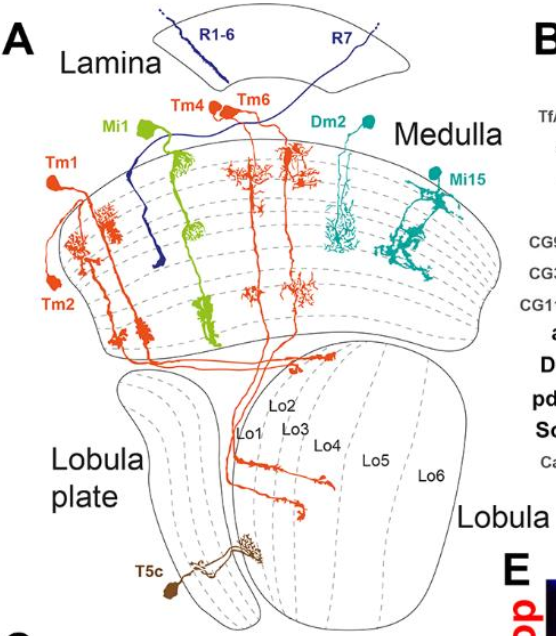
图1 黑腹果蝇视神经叶横截面
初步的神经元不同编码方式主要由于三种方式组成,一个是8个在不同区域转录因子以及信号分子组成,另一个是11个顺序表达时间的转录因子(temporal TFs)以及在姊妹神经元中表达的Notch信号。为了探究转录因子在发育过程中转录因子编码的方式,作者们希望寻找备选终端选择因子(Terminal selectors)的排列组合方式。174个神经元所组成的不同群组中,作者们在排除了泛神经以及广泛表达的基因找出了一系列转录因子。每个神经元群组中平均具有10个转录因子表达,所有的基因都被报道与视神经叶神经元种类身份表达相关。
Tm(Transmedullary)神经元1、2、4以及定义为神经元群组#62具有非常相近的转录谱,说明发育上的关系相近。作者们将#62的神经元群组定义为Tm6神经元。所有的Tm神经元具有相似的形态,但是成年期后深入到不同的小叶层之中(图1)。通过分析所有的终端选择因子,作者们发现这四个神经元群组共享相似的编码规则:ap、TfAP-2、scro、erm以及ct均表达在这四个神经元群组中。另外,这四组神经元又具有各自特异性的标记物,比如Drgx特异性表达在Tm1中,pdm3特异性表达在Tm2中,SoxN表达在Tm6中而Tm4中没有特异性表达的其他标记物。因此,这将是一个完美的神经元多样性命运解码模型。
Pdm3是唯一将Tm2与Tm4区分开的转录因子,作者们预测该终端选择因子如果缺失可能会导致神经元从Tm2转为Tm4,通过直到蛹期P50所有Tm神经元谱系表达但成年期只在Tm2特异性表达的Tmx/Tm2-Gal4品系,作者们产生了pdm3的突变克隆,从而确认Pdm3会作为Tm2转为Tm4类似神经元的功能。进一步地,通过Tmx/Tm2-Gal4特异性过表达Pdm3表达则会导致Tm4以及Tm6转变为Tm2,并同时伴随着细胞死亡的增加作为代偿反应。
与此类似的是,Drgx是唯一能够区分Tm1以及Tm4的转录因子。作者们使用直到蛹期P50表达在Tm神经元、成年期只在Tm4以及Tm6神经元中表达R35H01-Gal4过表达Drgx,发现Tm6仍然保持正常,但是Tm4会转变为Tm1。因此,Drgx负责特化Tm1的细胞命运。进一步地,通过染色质可及性分析寻找上游基因,作者们发现Drgx的增强子区域具有时间转录因子Klumpfuss(Klu)的结合位点。进一步地通过过表达Klu,作者们发现会增加Drgx+神经元的数量,证实了Klumpfuss调节Drgx的神经元编码选择的作用。
通过基因表达与功能分析,作者们发现一方面RTK信号通路负责稳定Tm选择器工作网络;另一方面,终端选择转录因子与蜕皮激素信号通路共同调节下游的靶标基因,从而形成了一个完整的神经元遗传学编码网络。
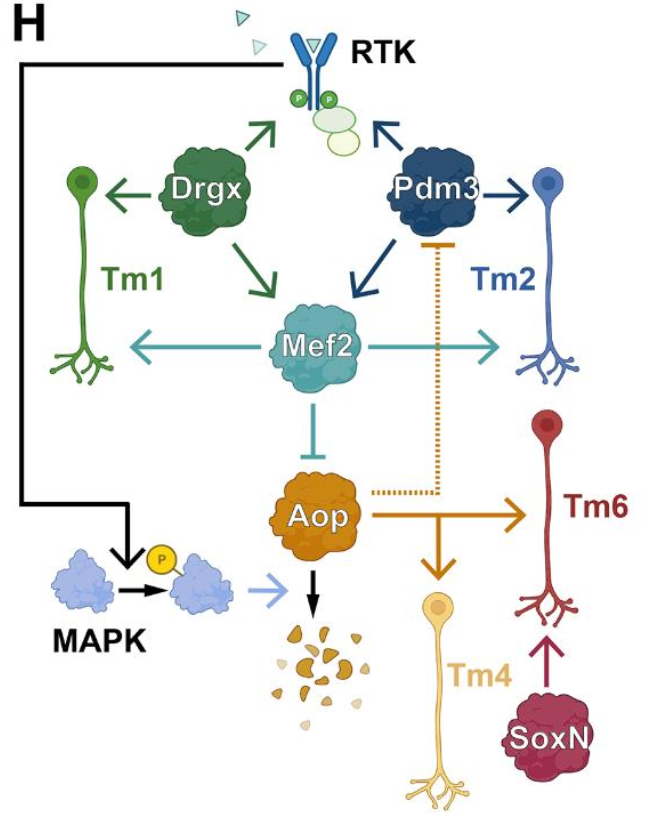
图2 工作模型
总的来说,作者们的工作通过视神经叶神经元组成确定了10个核心转录因子决定的神经元多样性决定网络,10个转录因子的排列组合加上时间转录因子以及蜕皮激素信号通路的配合,形成了神经元命运终端选择因子机制。
原文链接:
http://doi.org/10.1126/science.add1884
参考文献
1. Özel MN, Simon F, Jafari S, et al. Neuronal diversity and convergence in a visual system developmental atlas. Nature. 2021;589(7840):88-95. doi:10.1038/s41586-020-2879-3


想了解更多精彩内容,快来关注BioArt生物艺术
| 留言与评论(共有 0 条评论) “” |