
撰文 | 十一月
CRISPR以及CRISPR相关基因(Cas)为原核生物提供了针对病毒以及其他可移动遗传元件的特异性保护【1】。由CRISPR生成成熟的CRISPR RNA crRNA与Cas蛋白形成干扰复合体,引导其与互补的入侵序列的降解。CRISPR-Cas系统分为两大类、六小类和多个子类型【2】。巨型噬菌体(Jumbo phages)数量丰富,具有大于200 kb基因组,有些在细菌内部形成蛋白质核,包裹着噬菌体DNA进行复制和转录【3】。巨型噬菌体mRNA需要输出到细菌细胞质进行翻译,细菌利用这种情况通过使用RNA靶向的CRISPR-Cas系统来抑制噬菌体感染。通常情况下,在与互补噬菌体RNA结合后,III型CRISPR-Cas干扰复合体被激活,并启动一个多步骤的抗噬菌体核酸酶活性途径,该类型的免疫存在于多种细菌之中。但是巨型噬菌体诱导III型免疫的机制还尚不清楚。
2022年11月16日,新西兰奥塔哥大学Peter C. Fineran研究组在Molecular Cell上发表了文章Type III CRISPR-Cas provides resistance against nucleus-forming jumbo phages via abortive infection,巨型噬菌体在感染期间形成核类似的盾牌保护它们的基因组,而III型辅助核酸酶NucC诱导顿挫感染(Abortive infection)促进细菌防御。

先前的研究曾经发现细菌产生巨型噬菌体抵抗需要辅助核酸酶NucC【4】,因此作者们想知道辅助核酸酶NucC是否会作为CRISPR-Cas免疫机制。沙雷氏菌的NucC包含250个氨基酸,具有内切酶活性。为了阐明其结构以及激活机制,作者们在大肠杆菌中过表达并纯化了NucC,进一步测定了其晶体结构。NucC蛋白形成六聚体与两个配体cA3结合,由两对紧密排列的三聚体组成,面对面配对且相反三聚体的活性位点靠近在一起(图1)。
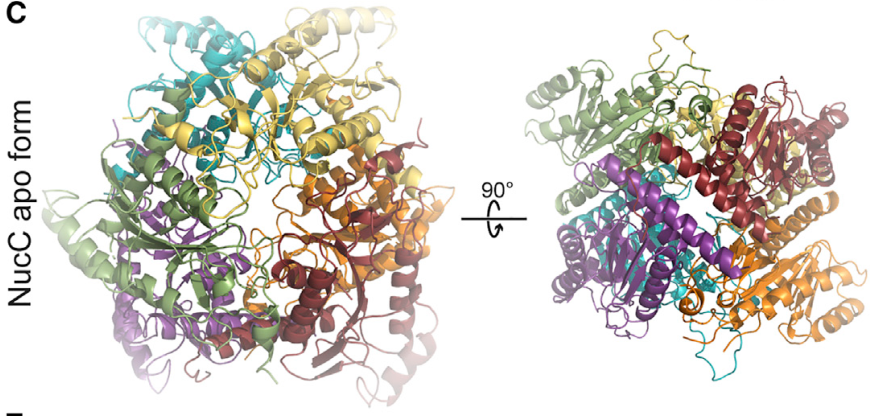
图1 NucC高分辨率晶体结构
鉴于对沙雷氏菌NucC辅助核酸酶活性的预测以及在巨型噬菌体免疫中可能的作用,作者们检测了其在体外存在cA3配体时降解核酸的能力。作者们发现在被cA3配体激活后,NucC降解双链DNA,此过程依赖于镁离子。NucC对DNA的降解依赖于NucC和cA3的浓度,预测的关键核酸酶活性位点残基D83N, E114N和K116L的突变会消除DNase的活性。另外,作者们发现NucC对超卷曲质粒DNA和线性PCR产物都具有DNA酶切活性。
进一步地,作者们在体内检测了NucC对于巨型噬菌体免疫的作用。作者们构建了NucC突变体细菌,突变体细菌的生长并不会都受到靶向或者非靶向CRISPR的影响。在质粒靶向诱导后,作者们对巨型噬菌体基因组以及质粒的完整性进行检测。在诱导后不到30分钟,基因组DNA被降解,质粒DNA从野生型宿主中被清除;相比之下,在没有NucC的情况下,基因组和质粒DNA的降解无法检测到。在III型系统的质粒RNA靶向作用下,激活的NucC降解细菌DNA,导致巨型噬菌体快速生长停滞以及细胞死亡。
作者们接下来研究了III型免疫和NucC对巨型噬菌体免疫机制作用,得到的实验结果表明,沙雷氏III-A型系统通过顿挫感染提供了对巨型噬菌体的免疫。顿挫感染指的是感染的细胞在噬菌体成熟和释放完成之前就已经死亡。III型免疫使野生型细胞中感染中心形成的效率降低了104倍;而在缺乏NucC的情况下,巨型噬菌体免疫大受影响,还存在部分的巨型噬菌体免疫可能是由于Cas7 RNase或Cas10 ssDNase活性的贡献。III型免疫对抗巨噬菌体感染是由NucC介导的细菌基因组降解提供的,进一步地作者们发现噬菌体DNA在核类似结构被保护着,免受NucC的切割。
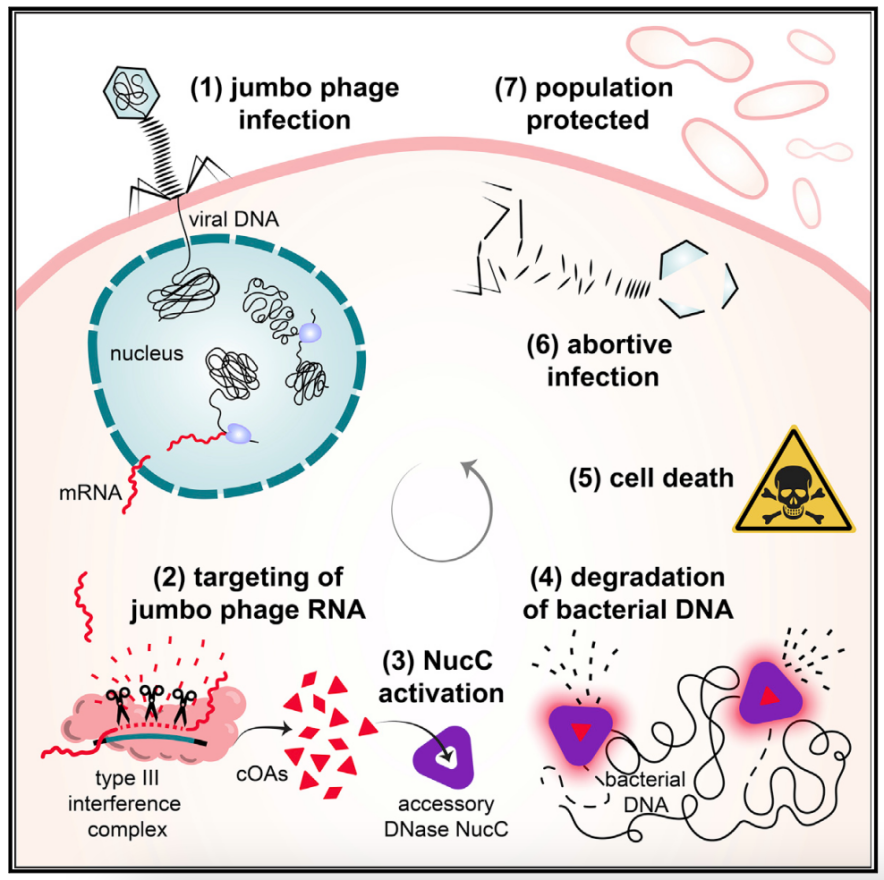
图2 工作模型
总的来说,作者们的工作发现III型CRISPR-Cas对巨型噬菌体的抵抗通过辅助核酸酶介导的顿挫感染在沙雷氏菌进行。巨噬噬菌体感染后被III型复合体识别,crRNA与病毒mRNA匹配,离开巨型噬菌体核进行翻译,巨型噬菌体mRNA的识别导致产生配体cA3,cA3次级信号被六聚体NucC蛋白结合,并促进DNase酶活性激活从而快速降解巨型噬菌体基因组DNA,实现细菌的免疫(图2)。
原文链接:
https://doi.org/10.1016/j.molcel.2022.10.028
参考文献
1. Barrangou, R., and Marraffini, L.A. (2014). CRISPR-Cas systems: prokaryotes upgrade to adaptive immunity. Mol. Cell54, 234–244. https://doi.org/ 10.1016/j.molcel.2014.03.011.
2. Makarova, K.S., Wolf, Y.I., Iranzo, J., Shmakov, S.A., Alkhnbashi, O.S., Brouns, S.J.J., Charpentier, E., Cheng, D., Haft, D.H., Horvath, P., et al. (2020). Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. Nat. Rev. Microbiol.18, 67–83. https://doi.org/10.1038/s41579-019-0299-x
3. Malone, L.M., Warring, S.L., Jackson, S.A., Warnecke, C., Gardner, P.P., Gumy, L.F., and Fineran, P.C. (2020). A jumbo phage that forms a nucleuslike structure evades CRISPR-Cas DNA targeting but is vulnerable to type III RNA-based immunity. Nat. Microbiol.5, 48–55. https://doi.org/10.1038/s41564-019-0612-5.
4. Malone, L.M., Warring, S.L., Jackson, S.A., Warnecke, C., Gardner, P.P., Gumy, L.F., and Fineran, P.C. (2020). A jumbo phage that forms a nucleuslike structure evades CRISPR-Cas DNA targeting but is vulnerable to type III RNA-based immunity. Nat. Microbiol.5, 48–55. https://doi.org/10.1038/s41564-019-0612-5


想了解更多精彩内容,快来关注BioArt生物艺术
| 留言与评论(共有 0 条评论) “” |